Моделирование днк что это
Ученые создали самую полную компьютерную модель гена ДНК
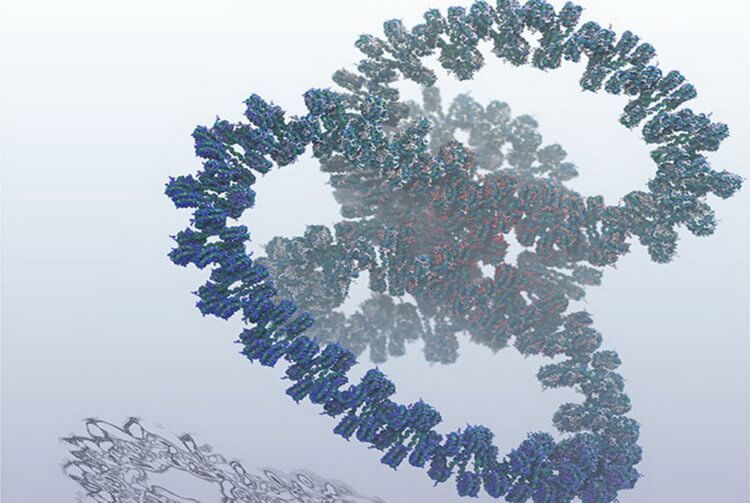
Ученые из Лос-Аламосской национальной лаборатории создали самую полную на сегодняшний день компьютерную модель всего гена ДНК, что потребовало моделирования одного миллиарда атомов. Специалисты отмечают, что их модель поможет исследователям лучше понять и разработать лекарства от таких болезней, как рак, сообщает пресс-релиз, опубликованный на портале Phys.org.
«Важно понимать ДНК на этом уровне детализации, потому что мы хотим точно знать, как гены включаются и выключаются. Знание того, как это происходит, может раскрыть секреты того, как развивается множество болезней», — комментирует Карисса Санбонмацу, структурный биолог из Лос-Аламосской национальной лаборатории.
Моделирование генов на атомистическом уровне является первым шагом к получению полного объяснения того, как ДНК расширяется и сжимается, что контролирует генетическое включение и выключение.
Биолог Карисса Санбонмацу и ее команда исследователей провели революционную симуляцию на суперкомпьютере Trinity в Лос-Аламосе, являющимся шестым по быстродействию в мире на сегодняшний день.
ДНК является основой для всего живого и содержит гены, которые кодируют структуры и деятельность человеческого организма. В человеческом теле достаточно ДНК, чтобы обернуть Землю 2,5 миллиона раз, что означает, что она уплотнена очень точно и организованно.
Длинная нитевидная молекула ДНК намотана на сеть крошечных молекулярных катушек. То, как эти катушки вращаются и раскручиваются, напрямую влияет на включение и выключение генов. Когда ДНК более компактна, гены выключаются, а когда ДНК расширяется, гены включаются. Исследователи пока не понимают, как и почему это происходит.
Хотя атомистическая модель является ключом к разгадке тайны, моделирование ДНК на этом уровне является непростой задачей и требует огромных вычислительных производительности.
«Прямо сейчас мы смогли смоделировать целый ген с помощью суперкомпьютера Trinity. В будущем мы сможем использовать суперкомпьютеры Exascale, которые дадут нам возможность смоделировать весь геном», — комментирует Анна Лаппала, физик из лаборатории в Лос-Аламосе.
Exascale — это новое поколение суперкомпьютеров, способных выполнять вычисления во много раз быстрее, чем современные машины. С такими огромными вычислительными возможностями исследователи смогут моделировать сразу весь человеческий геном, предоставляя еще больше информации о том, как гены включаются и выключаются.
В новом исследовании, опубликованном в издании Journal of Computational Chemistry, команда ученых из Лос-Аламоса объединилась с исследователями из японского Центра вычислительных наук RIKEN, учеными из Нью-Мексико и Университета Нью-Йорка, чтобы собрать большое количество различных экспериментальных данных и соединить их вместе, чтобы создать полностью атомную модель, которая согласуется с этими данными.
Компьютерное моделирование такого рода основано на экспериментах, включая захват конформации хроматина, криоэлектронную микроскопию и рентгеновскую кристаллографию, а также ряд сложных алгоритмов компьютерного моделирования, созданными специалистами Джейвун Ёнгом из RIKEN и Чань-Шун Тангом из Лос-Аламоса.
Обсудить новость можно в нашем Telegram-чате.
ДНК-оригами: путь от гравюры до нанороботов длиной в 30 лет
Журавлик из бумаги — один из самых известных образов в оригами
Автор
Редакторы
Сложить журавлика из бумаги — легко! Сложить журавлика из молекулы ДНК. тоже легко! Немного усидчивости и мастерства позволяют своими руками создавать из бумаги настоящие произведения искусства. Молекулы ДНК, в свою очередь, не требуют специальных навыков и собираются в красивые структуры на подобие оригами легко и непринужденно! Звучит как бред сумасшедшего, скажете вы. Отнюдь! Из этой статьи вы узнаете, как создать свою собственную фигурку оригами из ДНК, как похитить золото с помощью роботов, и кто победит в схватке между тараканом и ДНК-машиной.
«Биология — наука 21 века»
Эта работа публикуется в рамках конкурса научно-популярных статей, проведенного на конференции «Биология — наука 21 века» в 2014 году.
ДНК-оригами и связанные с этим ДНК-нанотехнологии сформировали в последнее десятилетие отдельное научное направление и получили стремительное развитие в работах нескольких научных групп по всему миру. В общем случае, за термином «ДНК-оригами» скрывается технология направленного конструировании молекул ДНК, способных к самосборке в заранее рассчитанные и смоделированные объекты. Такие конструкции могут быть как плоскими, так и объемными, довольно простыми и чрезвычайно замысловатыми. Все так же, как в японском искусстве складывания бумажного листа, только здесь вместо листа бумаги выступает нить ДНК!
Словарик
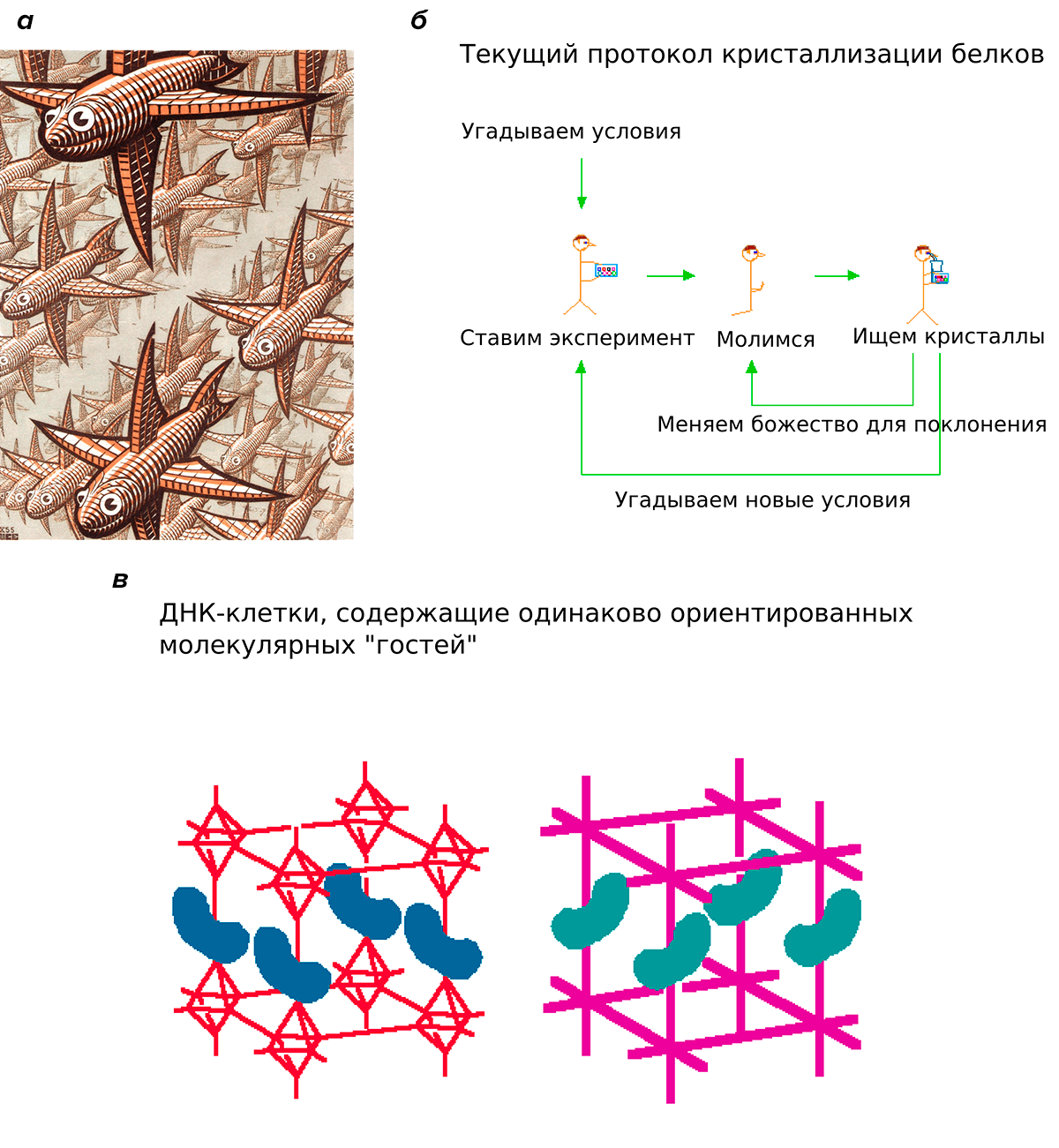
Как и многие научные открытия и разработки, это направление возникло, в некоторым смысле, случайно и неожиданно. Впервые о конструировании и использовании 3D-структур из ДНК всерьез заговорил американский ученый Нэд Симан (Ned Seeman) в начале 1980-х гг. Исследователь указывал на одну из главных сложностей метода рентгеновской кристаллографии (используемого тогда и по сей день для определения структуры белковых молекул), а именно необходимость подбора точных условий для получения «чистого» кристалла, по которым можно судить о структуре белка, и ставил своей целью разработку вспомогательной технологии фиксации белковых образцов (рис. 1). Для решения поставленных задач нужно было для начала разобраться с тем, как по собственному желанию и разумению собирать молекулы ДНК в необходимые конструкции.
Рисунок 1. а — Гравюра на дереве «Глубина», созданная Маурицем Корнелисом Эшером в 1955 году. Поговаривают, что, глядя на это произведение искусства в университетской столовой, Нэд Симан вдохновился на создание новой технологии, упрощающей кристаллизацию полипептидов и, следовательно, структурные исследования белков. С определением пространственной организации белков что-то не заладилось, но зато идеи Симана были подхвачены другими исследователями и привели к возникновению ДНК-оригами [1], [2]. б — Схема процесса кристаллизации белков, нарисованная Симаном. в — Идея ДНК-структур для правильной ориентации молекул в пространстве, изображенная Симаном.
Поиск и описание различных свойств элементарных ДНК-конструктов длились несколько лет. В 1991 году Нэд Симан представил нанометровый куб, ребра которого представляли собой молекулы ДНК [3]. Спустя некоторое время, несмотря на скептическое отношение некоторых ученых, работа была признана выдающейся. За неё Нэд Симан был удостоен Фейнмановской премии по нанотехнологиям в 1995 году и навсегда вошел в историю науки как создатель первых ДНК-нанотехнологий.
Результаты Нэда Симана и его лаборатории послужили фундаментом для идей другого блистательного исследователя и, без преувеличения, крупной фигуры в области ДНК-оригами — американца Пола Ротемунда. В 2006 году он опубликовал статью в авторитетнейшем научном издании Nature [4], в которой был описан метод получения точных ДНК-структур с заданной формой, а также были представлены детальные результаты и анализ такого направленного конструирования. В отличие от других исследователей, ему удалось строить не решетки из отдельных молекул, а настоящие плоские фигуры шириной в несколько цепочек ДНК (рис. 2). Эта статья сразу разлетелась по научно-популярным журналам, новостям и блогам, ведь представленные структуры и изображения впечатляли даже неподготовленного с научной точки зрения читателя. Не удивительно, что иллюстрации эксперимента красовались на обложке выпуска журнала.
Рисунок 2. Некоторые структуры, построенные при помощи ДНК-оригами и представленные в статье Пола Ротемунда
В последующие годы вышло несколько десятков статей, посвященных технологии ДНК-оригами. Росло число полученных форм, размеров конструкций и их сложности. Некоторые из результатов были экспериментально опробованы на реальных биологических объектах для решения прикладных биотехнологических и медицинских задач.
Двумерное ДНК-оригами: от простого к сложному
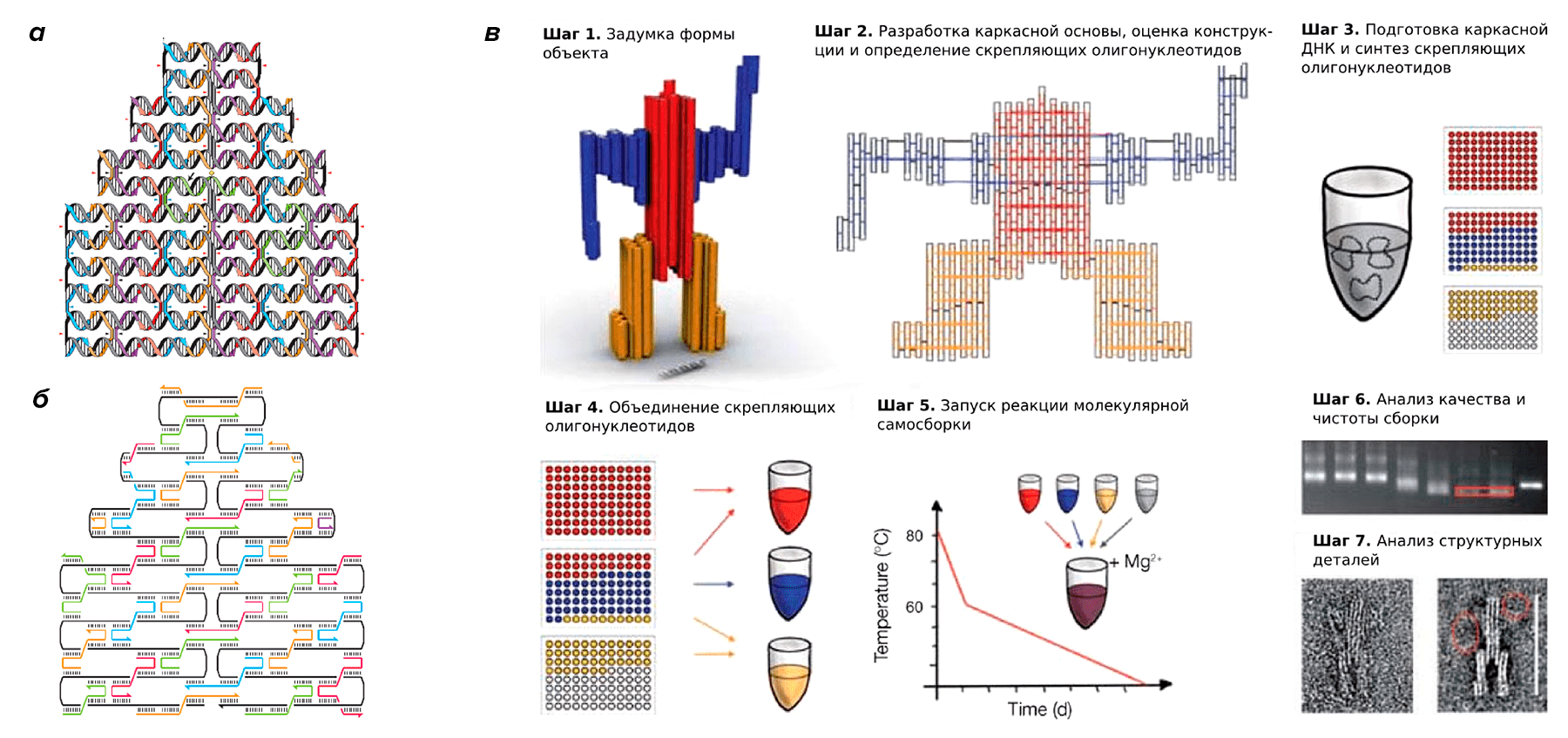
Как же ученые складывают ДНК-оригами? Разберемся в деталях данного метода. Для начала нам потребуется длинная одноцепочечная молекула ДНК, которая будет играть роль каркаса и основы нашего будущего объекта. В первых экспериментах использовалась ДНК фага M13 длиной 7249 нуклеотидов, однако сейчас с усовершенствованием ряда технологий стали использовать другие последовательности ДНК. Затем нам понадобятся заранее синтезированные короткие комплементарные цепочки ДНК (также называемые «скрепляющими цепочками» или «ДНК-скрепками», обычно 30-40 нуклеотидов в длину), последовательность которых необходимо подобрать при помощи компьютерного моделирования и анализа структур. Теперь смешаем растворы с длинной молекулой и короткими «скрепками» и нагреем смесь до температуры 95 °C, чтобы случайные и ненужные молекулярные связи распались. В процессе остывания до комнатной температуры (эта процедура называется отжигом) молекулы ДНК сами соберутся вместе, образуя нужную нам структуру. Проще простого — они всё делают за нас сами!
Рисунок 3. а, б — Иллюстрируют схему связей между каркасной ДНК (серая кривая) и скрепляющими олигонуклеотидами (кривые разных цветов). в — Пошаговая схема по изготовлению ДНК-оригами.
В результате эксперимента получается раствор, содержащий желаемые ДНК-конструкции. В одной-единственной капле раствора скрываются миллиарды крошечных объектов, которые, в отличие от бумажных фигурок оригами, нельзя потрогать, повертеть в руках и рассмотреть. Для оценки результата нам потребуется прибор со сверхвысоким разрешением — атомно-силовой микроскоп (АСМ) [6] или электронный микроскоп. Ведь рассмотреть фигурки размером 50-100 нм так непросто!
Для создания плоских структур ДНК-оригами смежные двухцепочечные молекулы должны быть соединены друг с другом кроссовером — особым типом переплетения нитей ДНК. Такое переплетение «склеивает» соседние цепочки посредством уотсон-криковского комплементарного спаривания и не дает всей структуре рассыпаться. Учитывая большое количество скрепляющих цепочек, требуются алгоритмы для расчета вероятности их точной посадки на основную цепь. Если ДНК-скрепка сядет не в том месте, то это может повлечь за собой как дефект структуры, так и полную путаницу в посадке всех остальных скрепок. В худшем случае это может привести к тому, что структура не соберется вовсе. Все-таки самосборка молекул в идеально плоскую структуру — это не такая уж и легкая задача.
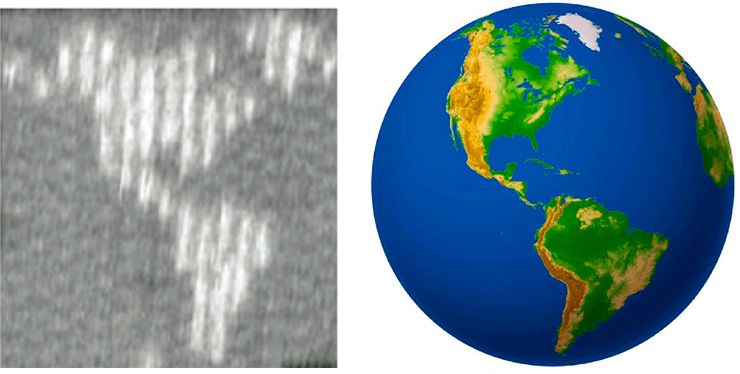
Рисунок 4. Точность собранного рисунка может быть довольно высока и находиться буквально на грани разрешения современных приборов. Можно добиться того, чтобы на ровном плоском «ДНК-полотне» в заранее предусмотренных местах будут выбиваться ДНК-шпильки. Это выглядит так, как если бы на кусочке ткани сделали рисунок узелками. Именно так была собрана карта западного полушария Земли, которую можно было увидеть исключительно при помощи АСМ.
Двумерные структуры на основе ДНК-оригами позволяют достичь не только большого многообразия форм — с помощью этой техники можно добиться невиданной до этого точности в размещении требуемых функциональных групп и молекул. Связанные с ДНК-скрепками молекулы могут быть размещены с точностью до нескольких нанометров и даже ангстрем (при условии правильной сборки)!
Если требуется собрать структуру побольше, нужно всего лишь соединить несколько длинных цепочек в одну составную конструкцию, как в конструкторе или крупных оригами-фигурах. На практике это можно осуществить так же, как было описано для одной единственной каркасной молекулы ДНК — нужно смешать все ингредиенты будущего объекта в одной пробирке, нагреть и ждать чуда, или собрать каждую деталь по отдельности, после чего объединить уже готовые элементы для окончательной сборки при менее интенсивном нагреве. В первом подходе нам приходится работать с достаточно большим количеством компонентов, ввиду чего увеличивается вероятность неправильной сборки молекул. При сборке деталей по отдельности необходимо провести несколько независимых экспериментов и совершить дополнительный шаг — повторный отжиг малых структур при нагреве до температуры 50 °C. При такой температуре детали еще не разваливаются на части, но уже более охотно связываются с друг с другом [7], [8].
Трехмерное ДНК-оригами
При определенных модификациях подход, который применяется для конструирования плоских структур, может быть обобщен до более сложного объемного случая. При конструировании 3D-структур можно, как и раньше, использовать кроссоверы, учитывая дополнительное третье измерение, и собирать все за один эксперимент, либо нужно начинать с собранных по отдельности плоских ДНК-объектов и лишь потом объединять их в конечную конструкцию. Выбор правильной последовательности действий в случае трехмерного ДНК-оригами чрезвычайно важен из-за значительно большего числа используемых молекул. Для особо сложных конструкций (особенно, при выборе первой стратегии сборки за один эксперимент) самосборка объекта может занимать несколько дней.
Несмотря на все сложности, которые могут возникнуть, объемные конструкции так привлекательны для исследователей! Ведь объемные объекты, ввиду многообразия возможных форм, могут быть использованы в широком круге самых разных прикладных задач.
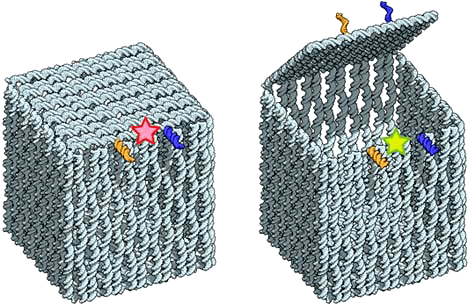
Так, используя несколько одинаковых квадратов, ученым удалось собрать полый куб [9] (правда, немного деформированный). Для устранения недостатков конструкции исследователи приделали к этому кубу крышку, которая запиралась на замок нанометровых размеров. Открытием крышки можно было управлять при помощи изменения конформации замка за счет спаривания с небольшими «ДНК-ключами» (рис. 5). Убедиться в том, что куб надежно закрывается на замок и открывается лишь определенным ключом, помог эффект FRET [17]. При этом данная конструкция стала одним из первых в своем роде контейнером для адресной доставки лекарств. Пока только в перспективе, конечно же.
Рисунок 5. ДНК-«коробочка» с открывающейся крышкой и молекулярным «замком». Получена в Датском центре ДНК-нанотехнологий в 2009 году. Предполагается, что в будущем такая конструкция будет использоваться для адресной доставки лекарств к определенным клеткам, где она будет открыта при помощи молекулярного «ключа».
Следующим этапом конструирования 3D объектов стала сборка строительных блоков, которые в дальнейшем скреплялись между собой, как детали конструктора (подробнее об этом можно прочесть в [10]).
Применение ДНК-оригами: ДНК-чипы, молекулярные машины и нанороботы
Пока мы затрагивали в основном процесс конструирования и сборки ДНК-оригами, и практически никак не упоминали о том, зачем все это нужно. И действительно, ведь ДНК-структуры разрабатываются не для того чтобы ими любоваться и получать эстетическое удовольствие! Современные ДНК-нанотехнологии направлены на решение нескольких прикладных задач, связанных с медициной, биотехнологией и программированием.
ДНК-конструкции могут нести на поверхности несколько строго ориентированных функциональных групп, специфически связывающих ту или иную молекулу, и, таким образом, регистрировать их присутствие. В самых простых случаях синтезируется специальная ДНК-скрепка с последовательностью, комплементарной молекуле РНК или ДНК в растворе. При использовании АСМ мы можем зафиксировать даже акт единичного связывания такой молекулы, так как при возникновении связи между структурой ДНК-оригами и целевой молекулой, последняя начинает сильно «выпирать» [11]. Это сразу бросается в глаза при анализе изображения.
Использование лигандов или аптамеров позволяет создавать настоящие сенсорные чипы. С их помощью можно регистрировать наличие не только одноцепочечных молекул нуклеиновых кислот, но и интересующих нас молекул белков и других соединений. При удачном стечении обстоятельств, речь может идти об обнаружении даже единичных молекул.
Способность к регистрации можно улучшить, фиксируя структуры ДНК-оригами на поверхности подложки. Подложка при этом заранее размечается методами литографии и травления, после чего обрабатывается специальными химическими соединениями. При правильной подготовке «плацдарма» для посадки, ДНК-структуры выстраиваются точно по порядку в интересующих нас местах и даже в нужной ориентации [12]. В совокупности, последовательность таких операций дает довольно точное размещение на подложке конструкций ДНК-оригами, которые, в свою очередь, служат подложкой для еще более точного размещения исследуемых молекул самой разной природы. Чип для широкого круга регистрируемых химических соединений готов к использованию!
Одним из интереснейших направлений ДНК-нанотехнологий является создание молекулярных машин, которые могли бы проводить разнообразные операции при минимальном участии человека. Например, Нэд Симан с коллегами собрал шагающую ДНК-машину с двумя ногами [13], [14]. На заранее сконструированной подложке (тоже собранной из ДНК) они разместили несколько других простых ДНК-машин, которые держали золотые наночастицы и могли их высвобождать при изменении конформации. Наш «молекулярный пешеход» ходил по подложке (по заранее известной дороге, которую тоже надо было собрать) и, когда оказывался вблизи носителей золота, отбирал у них золотую наночастицу! Заполучив немного золота, наш герой не успокаивался и шел за следующей порцией золотой добычи. По окончанию экспериментов жадный ДНК-пешеход должен был неплохо обогатиться!
Для того, чтобы продемонстрировать возможности программируемого перемещения молекулярных машин, другая группа исследователей собрала ДНК-«паука» с тремя ногами и одним хвостом [15]. (Странный, конечно, паук получился, но мы закроем на это глаза.) К ногам ДНК-«паука» были прикреплены функциональные молекулярные группы, которые позволяли перемещаться по специально созданной для этого трассе. Паук был привязан молекулой-замком за хвост в самом начале своего пути; затем, после связывания молекулы-замка с молекулой-ключом, его отпускали на свободу, и он убегал исследовать мир! Передвижение ДНК-паука было заснято в реальном времени при помощи микроскопии полного внутреннего отражения — его средняя скорость составила 3 нм/мин. Видимо, он не убегал, а скорее с наслаждением прогуливался по своей дорожке.
Большие надежды возлагаются на ДНК-оригами и другие ДНК-нанотехнологии в связи с вопросом адресной доставки лекарственных средств нуждающимся клеткам. К сожалению, это направление не проработано так хорошо, как другие, и всё ещё находится на стадии интенсивных исследований. Остается верить, что открытия, связанные с ДНК-роботами, служащими на благо здравоохранения и человечества в целом, ещё впереди!
Вместо заключения
К настоящему моменту учеными из разных стран собран большой объем экспериментальных данных и описано большое число механизмов на основе ДНК-технологий, которые ещё только предстоит полностью осмыслить и оценить. Уже сейчас подробно описать каждую из полученных структур и её преимущества над другими не представляется возможным. Ведь если только 10 лет назад исследованиями такого рода занималось всего несколько лабораторий во всем мире, сейчас их количество исчисляется несколькими десятками. Относительно будущего данной области науки сказать определенно можно только одно — дальше будет еще интереснее! Чтобы убедить вас в этом, приведем заголовок статьи, которая вышла в апреле 2014 года — Universal computing by DNA origami robots in a living animal, в которой описано использование ДНК-нанороботов в живых тараканах [16]. Уверяем вас, удивительное будущее не за горами!
СОДЕРЖАНИЕ
История
С самых ранних этапов структурных исследований ДНК с помощью дифракции рентгеновских лучей и биохимических методов, молекулярные модели, такие как модель двойной спирали нуклеиновой кислоты Уотсона-Крика, были успешно использованы для решения « загадки » структуры ДНК, а также для выяснения того, как последнее относится к его ключевым функциям в живых клетках. О первых высококачественных рентгеновских дифрактограммах A-ДНК сообщили Розалинд Франклин и Раймонд Гослинг в 1953 году. Розалинда Франклин сделала критическое наблюдение, что ДНК существует в двух различных формах, A и B, и дала наиболее четкие изображения обеих форм. Рентгеновская дифрактометрия. Первые расчеты преобразования Фурье атомной спирали были сообщены годом ранее Кокраном, Криком и Вандом, а в 1953 году за ними последовало вычисление преобразования Фурье спиральной катушки Криком.
Структурная информация генерируется из рентгеноструктурных исследований ориентированных волокон ДНК с помощью молекулярных моделей ДНК, которые сочетаются с кристаллографическим и математическим анализом рентгенограмм.
Первые сообщения о двойной спиральной молекулярной модели структуры В-ДНК были сделаны Джеймсом Уотсоном и Фрэнсисом Криком в 1953 году В том же году Морис Ф. Уилкинс, А. Стокса и HR Wilson, сообщает первые рентгеновские модели в vivo B-ДНК в частично ориентированных головках сперматозоидов лосося.
Важность
Давняя постоянная динамическая проблема заключается в том, как «самовоспроизведение» ДНК происходит в живых клетках, что должно включать временное разворачивание сверхспиральных волокон ДНК. Хотя ДНК состоит из относительно жестких, очень больших удлиненных биополимерных молекул, называемых волокнами или цепями (которые состоят из повторяющихся нуклеотидных единиц четырех основных типов, прикрепленных к дезоксирибозным и фосфатным группам), ее молекулярная структура in vivo претерпевает динамические изменения конфигурации, которые включают динамически прикрепленные молекулы и ионы воды. Суперспирализация, упаковка гистонами в хромосомных структурах и другие подобные супрамолекулярные аспекты также связаны с топологией ДНК in vivo, которая даже более сложна, чем молекулярная геометрия ДНК, что превращает молекулярное моделирование ДНК в особенно сложную проблему как для молекулярных биологов, так и для биотехнологов. Подобно другим большим молекулам и биополимерам, ДНК часто существует в нескольких стабильных геометриях (то есть проявляет конформационную изомерию ) и конфигурационных, квантовых состояниях, которые близки друг к другу по энергии на поверхности потенциальной энергии молекулы ДНК.
Такая изменяющаяся молекулярная геометрия также может быть вычислена, по крайней мере в принципе, с использованием методов квантовой химии ab initio, которые могут обеспечить высокую точность для небольших молекул, хотя недавно были сделаны заявления о том, что приемлемая точность может быть достигнута также для полинуклеотидов и конформаций ДНК. основа колебательного кругового дихроизма (ВКД) спектральных данных. Такая квантовая геометрия определяет важный класс неэмпирических молекулярных моделей ДНК, исследования которых еще только начались, особенно в отношении результатов, полученных с помощью VCD в растворах. Более подробные сравнения с такими неэмпирическими квантовыми вычислениями, в принципе, можно получить с помощью 2D-FT ЯМР-спектроскопии и исследований релаксации полинуклеотидных растворов или специально меченой ДНК, например, дейтериевыми метками.
Язык программирования: «ДНК»
Язык программирования: «ДНК»
От строчек кода до программируемых роботов!
Автор
Редакторы
Статья на конкурс «био/мол/текст»: Точная последовательность нуклеотидов в цепях РНК и ДНК, единственная ошибка в которой может привести к отказу в работе всей молекулы, невольно заставляет задуматься о схожести устройства нуклеиновых кислот с компьютерной программой, которая может «вылететь», если в ее коде всего в одном месте проставлена неверная буква. Данная статья посвящена необычным функциям, которые могут выполнять нуклеиновые кислоты. Это рассказ о различных молекулярных механизмах, логических схемах и даже роботах, созданных на основе ДНК, на которые мы предлагаем взглянуть почти как на блок-схемы и строчки программного кода.
Конкурс «био/мол/текст»-2018
Эта работа опубликована в номинации «Свободная тема» конкурса «био/мол/текст»-2018.
Генеральный спонсор конкурса — компания «Диаэм»: крупнейший поставщик оборудования, реагентов и расходных материалов для биологических исследований и производств.
Спонсором приза зрительских симпатий выступил медико-генетический центр Genotek.
В основу базовых знаний человека о биохимии его организма положена центральная догма молекулярной биологии, которая гласит, что переход генетической информации осуществляется последовательно, в одном направлении: от ДНК к РНК, а затем от РНК к белку. Разумеется, из этой догмы существуют свои исключения, как, например, подробно описанные на сайте ретровирусное направление передачи от РНК к ДНК, или прионное направление белок—белок. Тем не менее для подавляющего большинства организмов, правило выполняется.
Однако понятие переноса информации является термином весьма обтекаемым, и в данной статье внимание уделяется тому, как можно интерпретировать данный термин на базе известных нам этапов выполнения некоторых биологических процессов, и какие достижения прогнозируются или уже сделаны на основании этих интерпретаций. Иными словами, давайте взглянем на мир ДНК и РНК с точки зрения синтетического биолога: как это работает и как это можно изменить таким образом, чтобы оно работало именно так, как мы того пожелаем [1], [2].
Рассмотрим в качестве примера трансляцию по матрице мРНК — процесс синтеза белка с учетом заложенной в РНК информации. Процесс укладывается в повторяющиеся друг за другом три стадии, а именно инициацию, элонгацию и терминацию, и представляет собой типичный многоуровневый цикл. Подобные циклы легко укладываются в алгоритмы, а соответственно, могут быть записаны на большинстве современных языков программирования.
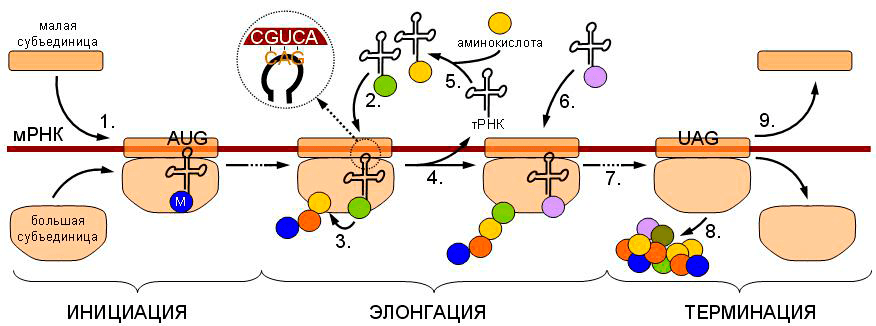
Например, для представленной на рисунке 1 схемы трансляции в примитивном виде можно записать следующее. ЕСЛИ рибосома встречает при движении вдоль мРНК стартовый кодон (триплет АУГ), ТО прикрепить к большой субъединице рибосомы аминокислоту метионин И затем НАЧАТЬ цикл. Сместить рибосому вдоль мРНК, И ЕСЛИ рибосома встречает при движении вдоль мРНК кодон, соответствующий определенной аминокислоте, ТО прикрепить к большой субъединице рибосомы эту аминокислоту И, ИНАЧЕ ЕСЛИ рибосома встречает при движении вдоль мРНК стоп-кодон, ТО открепить цепь белка от рибосомы и ЗАВЕРШИТЬ цикл.
Рисунок 1. Схематичное изображение процесса трансляции. Рибосома (две ее субъединицы изображены на картинке) движется вдоль нити мРНК, и, когда встречает на своем пути стартовый кодон метионина, воспринимает его как сигнал к началу цикла. Цикл заключается в наращивании белковой цепочки вплоть до момента, когда встречается стоп-кодон (УАГ на рисунке), воспринимаемый рибосомой как сигнал к завершению цикла.
Таким образом, операция, схематично представленная на рисунке 1, описывается простейшими операторами, присутствующими практически в любом языке программирования. Задавшись целью наиболее точно и верно описать весь процесс трансляции, при написании кода можно учесть в качестве начала цикла не стартовый кодон, а последовательность Шайна-Дальгарно, или Козак, или 5′-кэп; в теле цикла будет необходимо подробно описать сравнение антикодона текущей тРНК с текущим кодоном мРНК; описать диссоциацию рибосомных субъединиц и что делать в случае, если стоп-кодон так и не повстречался; задать возможность прохождения по одной мРНК нескольких рибосом одновременно — на самом деле, пунктов очень большое количество, однако все они могут быть включены в код программы посредством вложенных циклов с условием.
С подобной же идеей можно «разложить» в программный код и другие явления: репликацию ДНК или транскрипцию, да и не только матричные процессы. Например, и процесс прикрепления аминокислот к тРНК, и расщепляющие функции малых интерферирующих РНК, и работа R-плазмид или систем CRISPR-Cas основаны на принципах начала выполнения процедуры при встрече на пути исполнителя необходимого набора нуклеотидов. Этапы процедуры, а также ее завершение, если она является сложной, также определяются исключительно информацией, заложенной в коде соответствующих РНК и ДНК.
Вообще говоря, работа упомянутых здесь систем — малых РНК или палиндромных повторов (тех самых CRISP) — полна самых удивительных алгоритмов, и подобные системы уже сами по себе вполне могут считаться готовыми небольшими программами на языке нуклеиновых кислот. Этим «программам», конечно же, стоило бы посвятить целую статью, но, к счастью, на «Биомолекуле» уже есть отличные материалы по данным темам: «Обо всех РНК на свете, больших и малых», «Просто о сложном: CRISPR/ Cas» и «CRISPR-системы: иммунизация прокариот» [3–5].
Иными словами, последовательность нуклеотидов в нуклеиновых кислотах, как и последовательности символов в языках программирования, складывается в определенные «слова», которые воспринимаются исполнителями как команды проделать ту или другую операцию. С такого ракурса буквенную запись нуклеотидов (А, Т (У), Г, Ц) можно рассматривать как программный код, команды которого написаны с помощью четырехбуквенного алфавита. А сами молекулы нуклеиновых кислот в таком случае являются физическим воплощением подобного языка — реально существующим языком программирования.
Если исполнителями виртуальных языков программирования являются ЭВМ, то исполнителями реального языка ДНК в большинстве случаев можно назвать ферменты. Тем не менее при более детальном взгляде можно обнаружить, что некоторые реакции, выполняемые по принципу узнавания последовательностей РНК и ДНК, катализируются самими же молекулами РНК и ДНК. В частности, речь идет о каталитической активности особых последовательностей РНК, называемых рибозимами. Благодаря их существованию, нуклеиновые кислоты являются не только реальным, но еще и способным к самоисполнению программным кодом.
Макросы
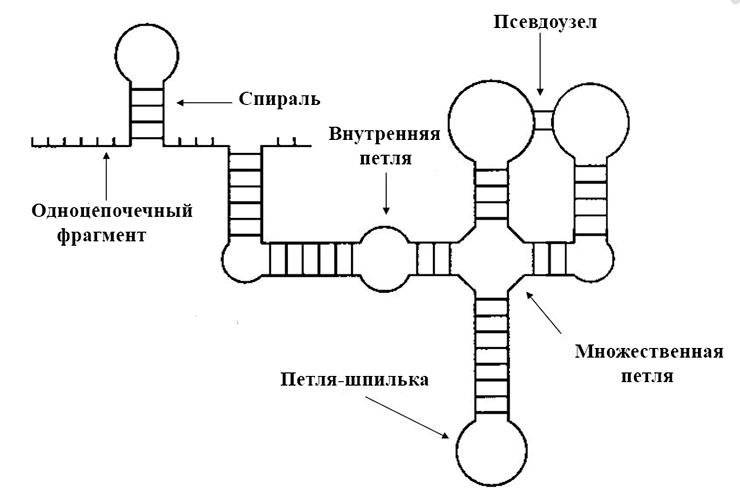
Главнейшую роль в исполнении команд, связанных с поиском и распознаванием соответствующих участков кода, играет известный принцип комплементарности. Однако и в двуцепочечной структуре ДНК, и, гораздо чаще, в структуре одноцепочечных и гибридизованных (соединившихся) РНК встречаются участки, на которых нуклеотиды не подходят друг другу для образования пары оснований. В таком случае образуются шпильки, петли, узлы и другие структуры (рис. 2). Иногда они не несут в себе никакого принципиального значения, но в других случаях их образование — причем в строго определенном месте и составленное из строго определенного набора нуклеотидов — является ключом для выполнения той или иной операции [6].
Рисунок 2. Схематичное изображение возможных образований во вторичной структуре одноцепочечных молекул нуклеиновых кислот
Операции, выполняемые подобными участками, могут быть самыми различными, и ниже мы поговорим о некоторых из них. Но перед тем укажем, что подобные структуры могут образовываться не только в пределах одной молекулы, но и при неполной комплементарности двух и более цепей РНК или ДНК [7]. Представим себе два коротких фрагмента ДНК: один вида 5′-ААГТАТЦ(X)ТАГЦЦГА и другой вида ТТЦАТАГАТЦГГЦТ-5′, где (X) — это какая-то недлинная последовательность нуклеотидов, которая как раз и будет выполнять некоторую операцию. Второй фрагмент ДНК комплементарен первому по краям от (X), и потому эти два фрагмента могут гибридизоваться друг с другом, образуя двуцепочечную систему, у которой посередине будет наблюдаться «выпячивание», благодаря неспаренному участку (X) первого фрагмента.
Таким образом, если (X) выполняет какую-то функцию — например, расщепляет второй фрагмент ДНК в строго определенном месте, — то весь фрагмент 5′-ААГТАТЦ(X)ТАГЦЦГА целиком выполняет уже две следующие друг за другом операции: распознавание строго определенной комплементарной ему последовательности и, если распознание выполнено успешно, ее расщепление. Итого, мы получаем фрагмент программного кода, независимый в исполнении своих функций от других элементов программы (от генетического кода организма или других фрагментов поблизости). Подобные структуры в среде программистов иногда называют макросами; воспользуемся этим термином и мы.
Рибозимы
Уже упоминаемые выше молекулы РНК, проявляющие ферментативные свойства и способные разрезать другие последовательности ДНК и РНК, являются именно такими «макросами». Рибозимы — это короткие последовательности РНК, условно состоящие из двух частей. Каталитический участок, последовательность которого строго предопределена, выполняет функцию расщепления другой молекулы ДНК или РНК в определенном сайте. Гибридизационный участок, последовательности которого могут быть различны, окружает каталитический с двух сторон, и отвечает за распознавание того участка другой молекулы ДНК или РНК, которую необходимо разрезать, и присоединение непосредственно к ней.
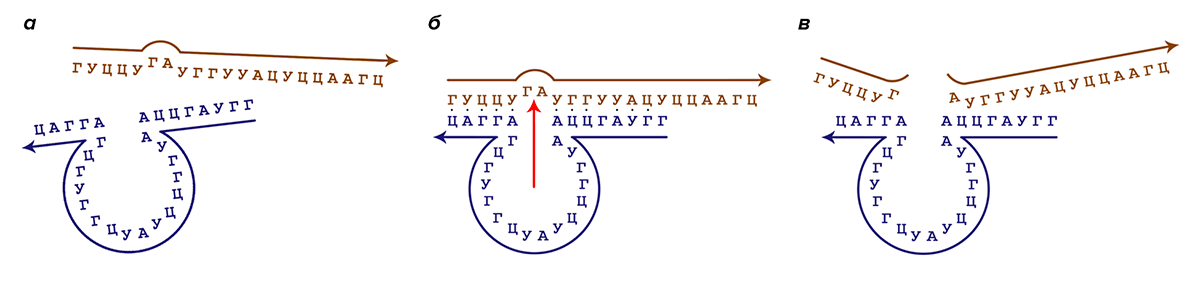
Схематично процесс работы рибозимного «макроса» представлен на рисунке 3.
Рисунок 3. Принцип работы рибозимов. Синим цветом выделена последовательность рибозимного «макроса», коричневым цветом — целевая для него последовательность РНК, которую он призван расщеплять. Стрелками указано направление от 5′- к 3′-концу молекул. а — Молекулы «макроса» и целевой РНК встречаются в растворе, происходит операция логического сравнения. б — Если последовательности комплементарны и сравнение дало положительный ответ, молекулы гибридизуются, и выполняется операция расщепления РНК в строго определенном месте (указано красной стрелкой) рибозимной частью «макроса». в — Фрагменты расщепленной молекулы РНК отщепляются от рибозимного «макроса».
Стоит отметить, что помимо рибозимов существуют и дезоксирибозимы — молекулы ДНК, также проявляющие каталитическую активность и при этом имеющие большую стабильность самих молекул [8], [9].
Аптамеры
По несколько иному принципу работают аптамерные «макросы». Нуклеиновые аптамеры — это короткие последовательности ДНК или РНК, которые способны распознавать присутствие других молекул (обычно сложных органических веществ) поблизости и, если рядом находится молекула определенного вещества, то захватывать ее, образуя кольцо или шпильку вокруг цели. Подобный захват может приводить к различным итогам [10]. В частности, некоторые органические вещества при аптамерном захвате немного изменяют свою пространственную структуру, что может приводить, например, к изменению их фотохимических свойств [11], [12].
Аптамерный участок цепи ДНК или РНК, продолженный участками, комплементарными к какой-либо целевой нуклеиновой кислоте, может служить способом обнаружения присутствия в системе этой кислоты. Таким образом, аптамерный «макрос» выполняет две последовательные функции: обнаружить целевую РНК или ДНК и затем подать сигнал в случае ее обнаружения.
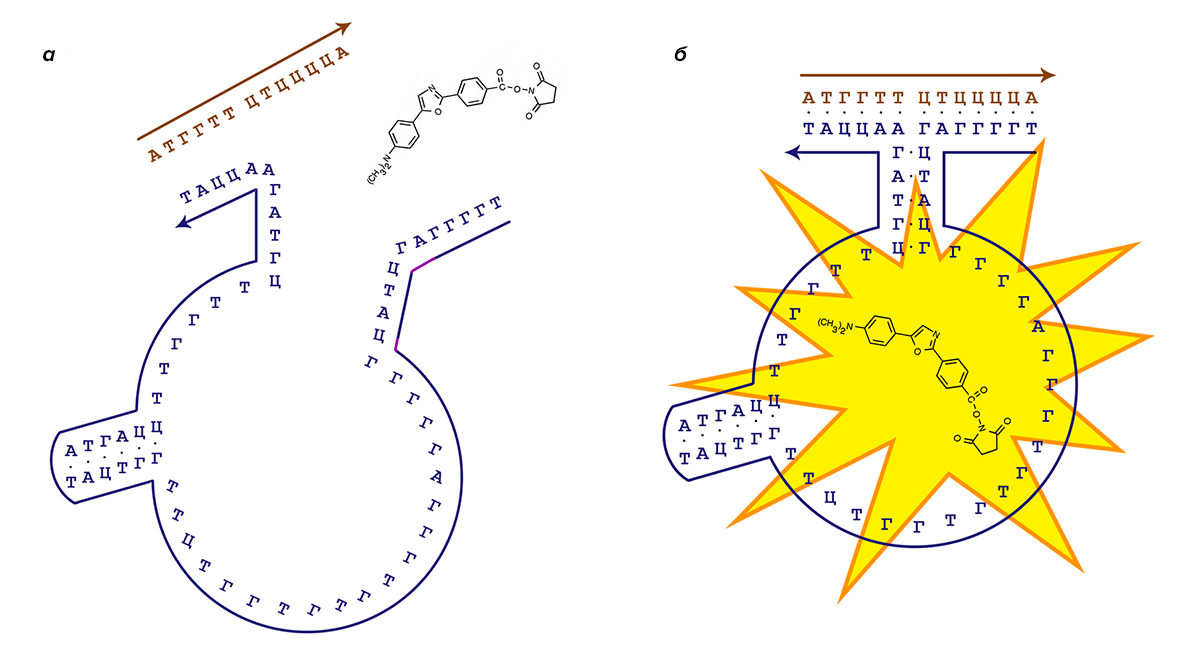
Подобные аптамерные конструкции могут использоваться в качестве сенсоров [13], [14], когда захват молекулы-флуорофора «макросом» происходит только в присутствии целевой последовательности ДНК. Схематично процесс работы подобной системы представлен на рисунке 4.
Рисунок 4. Принцип работы аптамерных конструкций. Фиолетовым цветом выделена последовательность аптамерного «макроса», коричневым цветом — целевая для него последовательность ДНК. а — В отсутствии целевой последовательности «макрос» не может соединиться в аптамерный замок из-за недостаточной стабильности такого замка. б — Целевая последовательность ДНК, будучи узнанной, стабилизирует замок, и захваченная «макросом» молекула (в данном случае это модифицированная молекула дапоксила) подает интенсивный флуоресцентный сигнал.
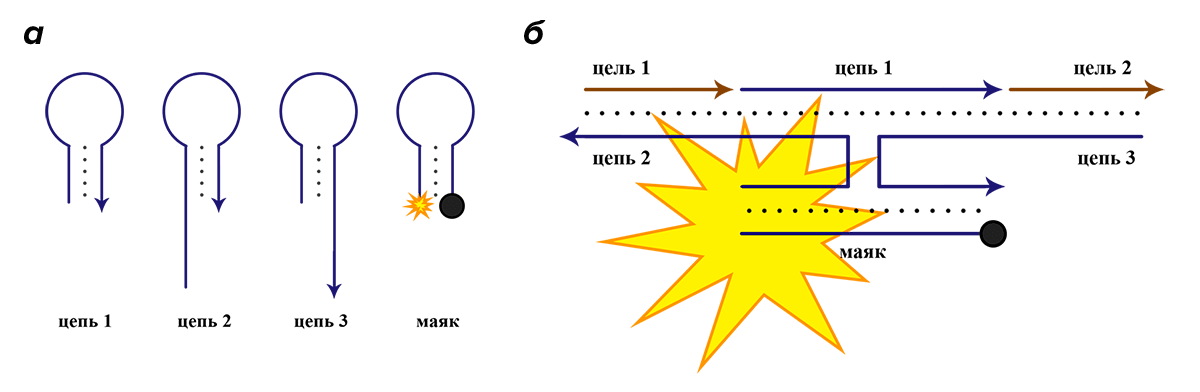
Молекулярные маяки
Испускание флуоресцентного сигнала «макросом» может программироваться не только путем аптамерного захвата какой-нибудь флуоресцирующей молекулы. Альтернативным вариантом является вид ДНК-зондов [15], называемый молекулярными маяками — короткие самогибридизующиеся фрагменты ДНК, меченные двумя веществами: флуорофором (соединением, способным испускать свет при облучении) и гасителем флуоресценции. Сам по себе такой фрагмент имеет вторичную структуру, при которой флуорофор и гаситель находятся рядом, вследствие чего весь сигнал флуорофора поглощается гасителем, и наблюдателю никакого сигнала не заметно. Однако если по близости от маяка присутствует другая молекула ДНК или РНК, комплементарная последовательности маяка, то маяк раскрывается и гибридизуется с ней, что ведет к удалению на значительное расстояние гасителя флуоресценции от флуорофора, и в результате наблюдатель видит сигнал, более не подавляемый гасителем.
Подобное явление гашения флуоресценции является частным случаем явления, называющегося FRET (резонансный перенос энергии по Ферстеру). Подробнее его принципы и причины описаны в соответствующей статье на сайте: «Рулетка для спектроскописта» [16].
Иными словами, данный «макрос» так же, как и аптамерный, выполняет две функции: обнаружение целевого фрагмента ДНК или РНК и информирование наблюдателя в случае его обнаружения. Схематично принцип его работы представлен на рисунке 5.
Рисунок 5. Принцип работы ДНК-зондов типа молекулярных маяков. Синим цветом выделена последовательность маякового «макроса», на одном из концов которой находится флуорофор (желтый), а на другом — гаситель флуоресценции (серый); коричневым цветом — целевая для него последовательность РНК. а — В отсутствии целевой последовательности «макрос» находится в состоянии шпильки, и из-за близости гасителя к флуорофору сигнала не наблюдается. б — При узнавании целевой последовательности ДНК и связывании с ней, шпилька раскрывается, флуорофор отдаляется от гасителя, и «макрос» подает интенсивный флуоресцентный сигнал.
Различные ДНК-зонды, в частности молекулярные маяки, уже много лет применяются в полимеразной цепной реакции в режиме реального времени (или количественной ПЦР), благодаря которой количество специфической последовательности ДНК в продукте можно отслеживать непосредственно в процессе самой реакции, не дожидаясь завершения последнего цикла и проведения какого-либо количественного анализа.
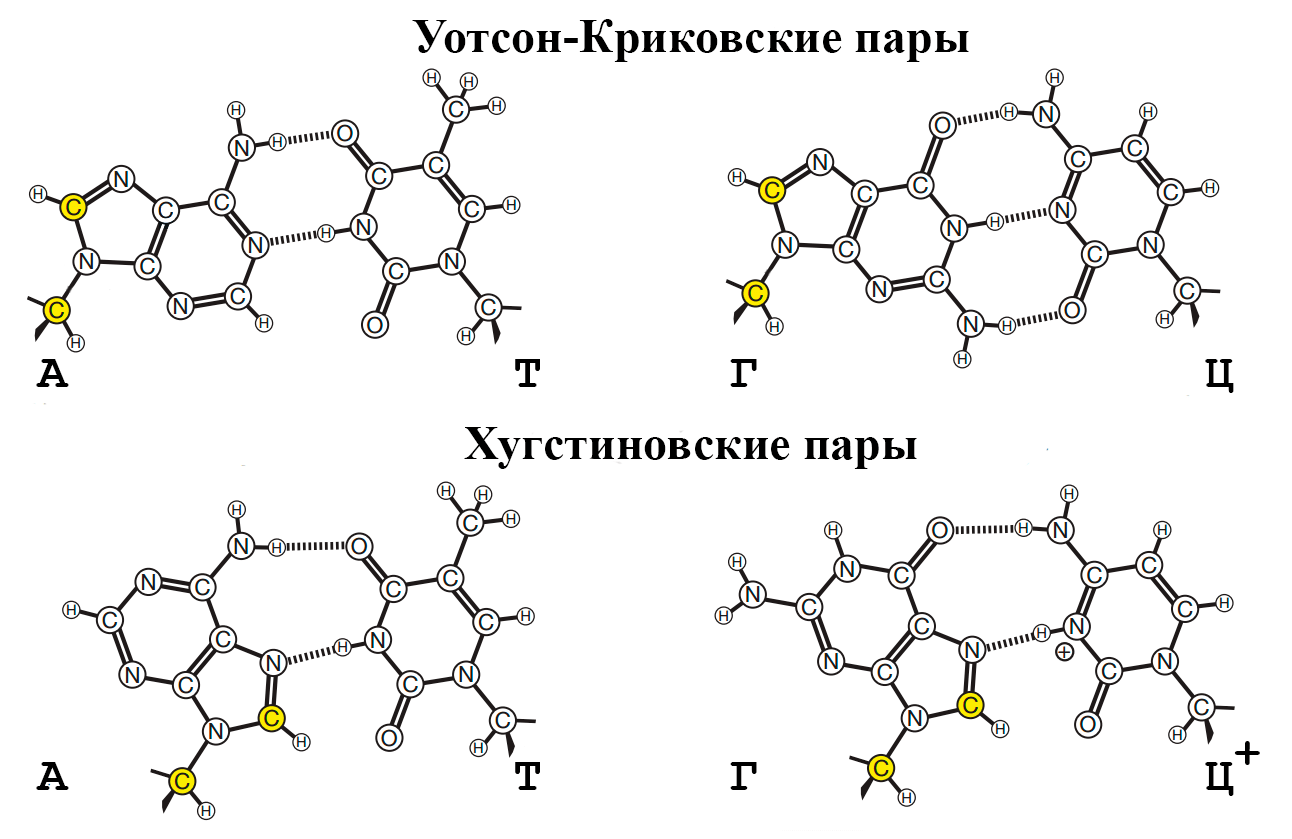
Хугстиновые носители
Нуклеотиды могут объединяться друг с другом не только по принципу комплементарности, названному так Джеймсом Уотсоном и Френсисом Криком, но и целым рядом других механизмов. Одним из таких механизмов является хугстиновское взаимодействие (по имени открывшего его Карста Хугстина), которое работает по схожему комплементарному взаимодействию, однако, выражаясь простым языком, нуклеотиды в составе нуклеиновых кислот при хугстиновском взаимодействии соединяются в направлении, значительно повернутом относительно уотсон-криковского (рис. 6).
Рисунок 6. Разница в образовании хугстиновской и уотсон-криковской комплементарных нуклеотидных пар. Штриховыми линиями обозначены водородные связи между атомами.
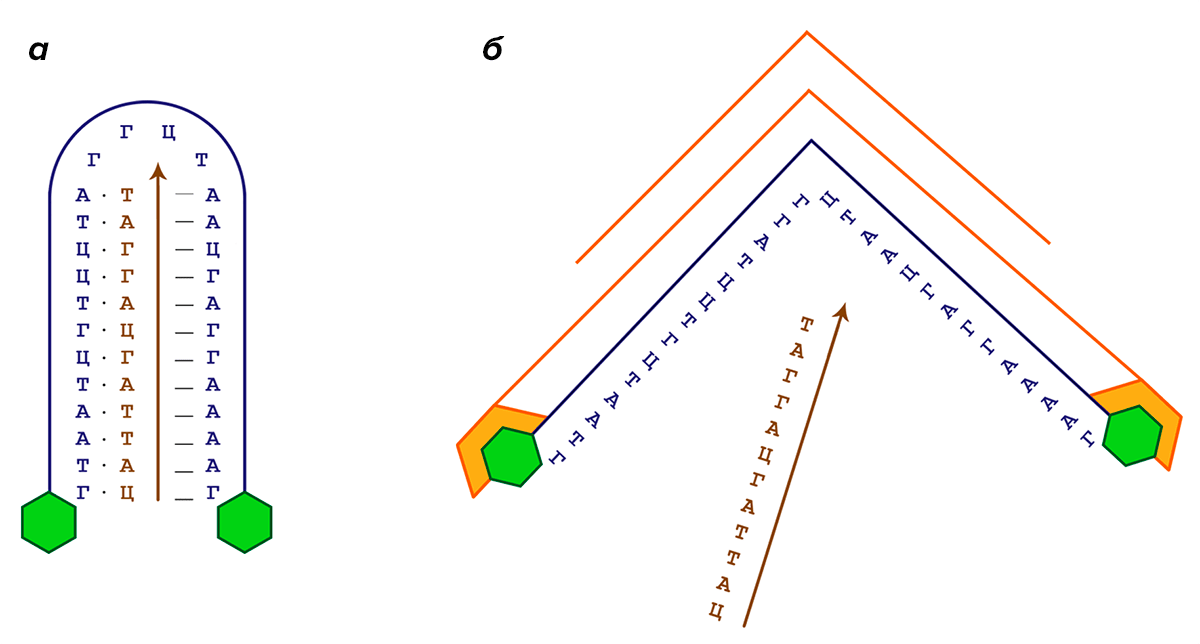
Таким образом, гипотетически один нуклеотид может вступать в комплементарное взаимодействие сразу с двумя другими нуклеотидами. На основе этой возможности ученые синтезировали одноцепочечную молекулу ДНК, один фрагмент которой был комплементарен целевой молекуле нуклеиновой кислоты по принципу Уотсона и Крика, а второй — по принципу Хугстина. В результате этого в присутствии целевой ДНК или РНК создавался комплекс, внутри которого мишень была сохранена от внешних воздействий и в то же время сама не проявляла никакой активности. Иными словами, синтезированная последовательность ДНК выполняла оператор с условием (нахождение рядом мишени) и, в случае положительного ответа, выключала функцию другой молекулы.
Вторым пунктом, которому должен соответствовать подобный «макрос», чтобы называться не замком, а носителем, является функция высвобождения целевого фрагмента именно в том месте организма, где от него требуется. Это условие можно запрограммировать, например, добавив на концы молекулы-носителя небольшие органические фрагменты, воспринимаемые в качестве антигенов различными иммунными тельцами нашего организма [17]. В среде, где присутствуют антитела конкретного типа, они свяжутся с молекулой-носителем и таким образом «раскроют» ее цепь и высвободят содержимое. Принцип работы такого носителя представлен на рисунке 7.
Рисунок 7. Принцип работы хугстиновского носителя на основе отношений антитело—антиген. Синим цветом выделена последовательность «макроса»-носителя, на обоих концах которого находятся молекулы антигена (зеленые); коричневым цветом — последовательность ДНК, переносимая «макросом». а — «Макрос» в связанном состоянии; точками отмечены уотсон-криковские пары, штрихами — хугстиновские. б — Антитело (оранжевое), специфическое к меткам на концах «макроса», связывается с носителем и раскрывает его, тем самым высвобождая содержимое.
Эти фрагменты по обоим концам могут быть одинаковыми или различными, и второй случай соответствует логическому сравнению: высвобождение будет происходить только тогда, когда вокруг молекулы-носителя имеется и один вид антител, и другой. Возможность встраивания функции сравнения повышает точность подобной адресной доставки.
На самом деле, разнообразие функций, которые могут исполнять различные последовательности ДНК, куда более обширны. Например, есть исследования по созданию «макросов»-детекторов, зависящих от светового [18–20] и радиоактивного излучений или наличия определенных молекул вокруг [21], или «макросов»-носителей, зависящих от электрических импульсов [20], [22].
Логические элементы
Принцип комплементарности, по которому связываются между собой нуклеиновые кислоты, является инструментом для создания практически идеально селективных (не допускающих ложного срабатывания) условий объединения двух молекул. Однако по такому же принципу могут объединяться в одну систему не только две молекулы, но и гораздо большее их число, если различные фрагменты этих молекул комплементарны другим фрагментам других молекул. Примером подобных систем служит неоднократно уже освещавшееся во многих научно-популярных источниках ДНК-оригами [23], в том числе и на «Биомолекуле»: «ДНК-оригами: путь от гравюры до нанороботов длиной в 30 лет» [24] и «Голактеко опасносте: ДНК-роботы в живом организме» [25].
Это создает отличные условия для моделирования на языке ДНК логических операций, гораздо более сложных, чем унарное «ДА». Используя описанные выше молекулярные маяки или другие ДНК-зонды со схожим механизмом, можно создавать системы, способные проводить более сложное логическое сравнение [26], [27]. Одной из таковых является логическая операция «И» (рис. 8), дающая положительный ответ только в случае присутствия двух различных сигналов [28]. Это может помочь в проведении комплексных исследований с участием нескольких целевых фрагментов ДНК или РНК, что очень важно для клинической диагностики многих заболеваний.
Рисунок 8. Идея логического элемента «И», написанного на языке ДНК. Синим цветом выделены последовательности, принадлежащие коду логического элемента. Коричневым цветом выделены целевые фрагменты ДНК или РНК, которые элемент призван обнаружить вместе. а — Логический элемент состоит из четырех коротких фрагментов ДНК, один из которых является молекулярным маяком. б — Только в присутствии обеих целевых последовательностей части логического элемента гибридизуются полностью и подается флуоресцентный сигнал. Для упрощения схемы нуклеотидный код каждой цепи не указан.
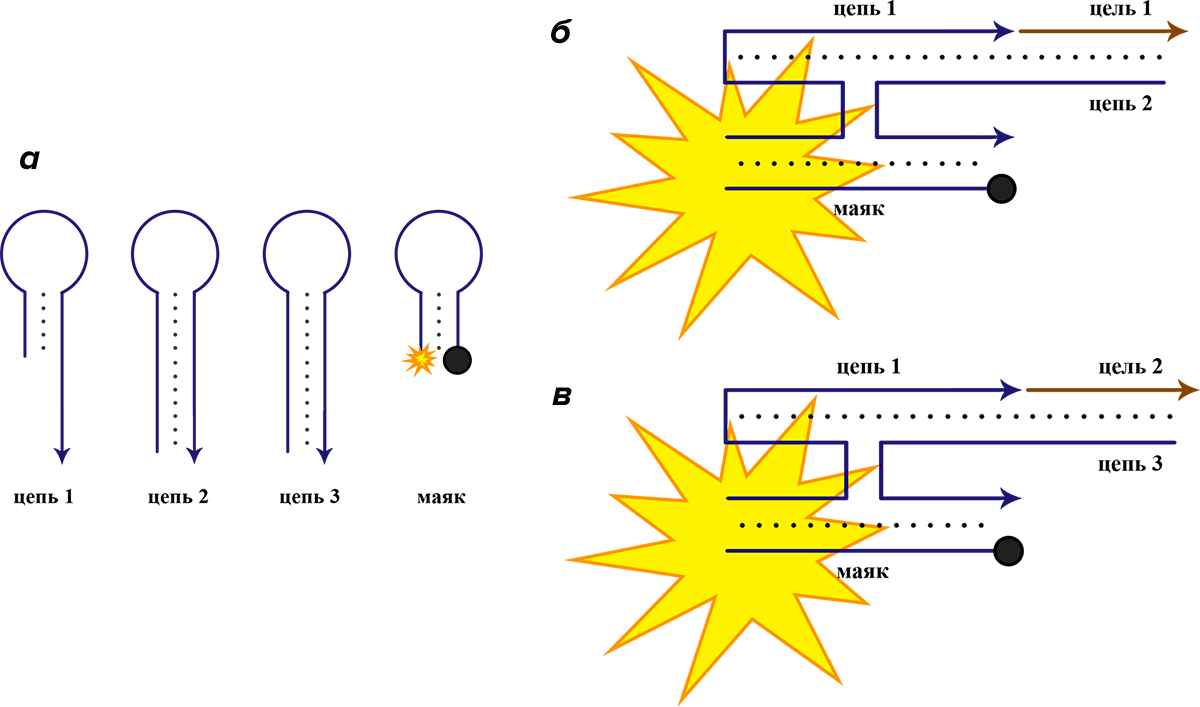
Напротив, можно создать конструкции «ИЛИ», которые будут давать положительный ответ и в присутствии только одной из двух (или даже более) целевых молекул нуклеиновых кислот (рис. 9). Или же конструкции типа «Исключающее ИЛИ», которые дают сигнал в присутствии только одной из целевых последовательностей, и не дают сигнал, когда присутствуют обе, что очень полезно при исследовании объектов с частыми мутациями в геноме — например, туберкулеза [29].
Рисунок 9. Идея логического элемента «ИЛИ», написанного на языке ДНК. Синим цветом выделены последовательности, принадлежащие коду логического элемента. Коричневым цветом выделены целевые фрагменты ДНК или РНК, которые элемент призван обнаруживать. а — Логический элемент состоит из четырех коротких фрагментов ДНК, один из которых является молекулярным маяком. б и в — В присутствии любой из целевых последовательностей три из четырех частей логического элемента, включая маяк, гибридизуются между собой, и подается флуоресцентный сигнал. Для упрощения схемы нуклеотидный код каждой цепи не указан.
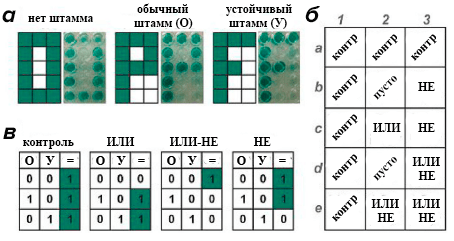
Простейшие «ДА»-элементы, положенные в основу теста на туберкулез и его тип (Xpert MTB/RIF), уже активно используются в ряде стран и пропагандируются Всемирной организацией здравоохранения. В это же время модернизированные логические конструкции, такие как элементы «ИЛИ», «НЕ» или «ИЛИ-НЕ» (он же стрелка Пирса) на основе ДНК позволяют значительно облегчить подобные тесты [30]. Пример такого применения представлен на рисунке 10.
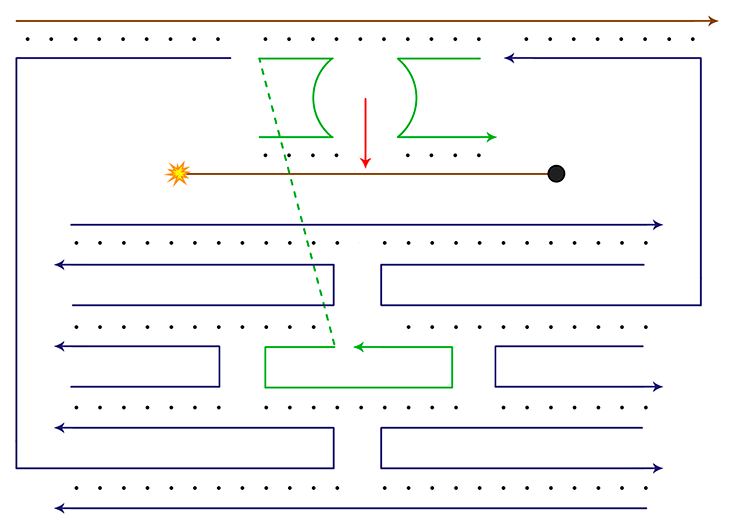
Рисунок 10. Идея экспресс-теста на определение рифампицинустойчивого штамма палочки Коха с помощью логических элементов на основе ДНК. В каждую лунку планшета вносится определенный логический элемент ДНК, а затем в каждую — одна и та же проба. а — Слева: теоретически ожидавшийся результат; справа: окраска лунок в зависимости от того, какая проба была добавлена на планшет. б — Схема нанесения логических элементов на планшет. в — Принцип окрашивания каждого логического элемента в зависимости от того, какая проба вносится в ту же лунку, где он находится.
Программируемые роботы
Итого, мы имеем язык программирования, записанный в реальности и имеющий возможность взаимодействовать с реальными объектами. В этом языке нам уже известна запись достаточно большого числа команд и операций и даже независимых групп команд («макросов»), которые выполняют широкий спектр функций. Более того, большинство из перечисленных выше «макросов» являются самоисполняемыми: ни рибозимы, ни аптамерные или маяковые сенсоры не нуждаются в стороннем ферментативном исполнении заложенных в них команд, как и логические элементы — достаточно простого удовлетворения команды-запуска «макроса» (в большинстве случаев это просто встреча с целевой последовательностью ДНК или РНК).
Однако проектирование и применение подобных макросов для повседневных задач, будь то детектирование бактериальных штаммов или же лечение каких-либо заболеваний, имеет ряд недостатков. Рассмотрим таковые, задавшись целью использовать дезоксирибозимный «макрос» для расщепления РНК вируса гриппа A, попавшего в клетку. Расщепление ее в определенном месте приведет к неспособности вируса воспроизводить копии себя и, как следствие, распространение вируса по организму прекратится, что будет означать, что, когда вся вирусная РНК окажется расщепленной, человек успешно выздоровеет.
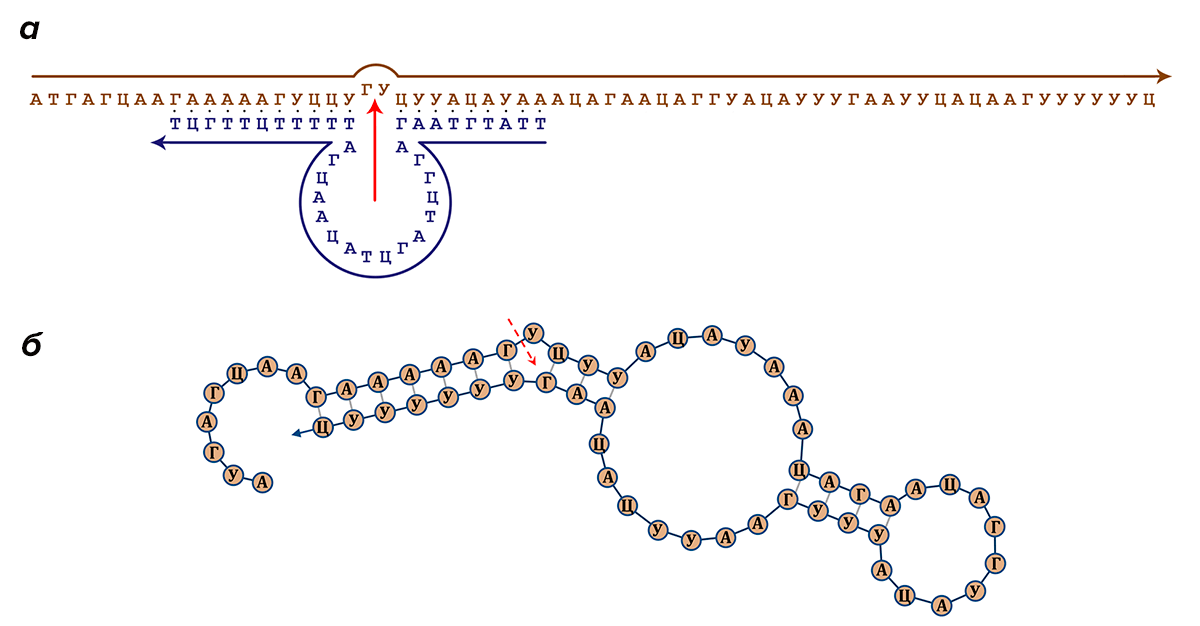
Возьмем короткий шестидесятинуклеотидный фрагмент вирусной РНК, в котором содержится участок, который мы хотим расщепить дезоксирибозимом (воспользуемся базой данных GenBank). Спроектируем ту часть «макроса», что отвечает за распознавание, таким образом, чтобы она была полностью комплементарна нуклеотидам, что окружают целевой участок (рис. 11а). Основу дезоксирибозима выберем из известного исследования [31].
Рисунок 11. Строение фрагмента РНК вируса гриппа A. а — Запись кода шестидесятинуклеотидного фрагмента вирусной РНК с «нацеленным» на него дезоксирибозимом для расщепления. б — Двумерная проекция вторичной структуры данного фрагмента, предсказанная программой MFold.
Однако программный код (а это фактически он), представленный на рисунке 11а, является лишь проекцией, виртуальной копией реального кода. Иными словами, необходимо чтобы «макрос» подходил к целевому фрагменту РНК не только с точки зрения логического сравнения, но и с точки зрения физических законов мира: как минимум, термодинамических и стереохимических, а также чтобы окружающая среда (ионный состав, ее кислотность) была оптимальна для работы «макроса».
На рис. 11б представлена плоская проекция вторичной структуры выбранного шестидесятинуклеотидного фрагмента. Как видно, доступ к целевому участку этого фрагмента в естественном состоянии гораздо более затруднен, чем это было изображено в строчке программного кода. Учитывая то, что каждая буква в подобном коде — это даже не массивный атом, а целая группа из двух десятков атомов, трудность подсоединения дезоксирибозимного макроса к подобной конструкции становится понятной даже с точки зрения бытовой логики. Как следствие, результат работы дезоксирибозима уже будет очень далек от стопроцентного.
Такими вопросами, связанными с реальной конфигурацией молекул нуклеиновых кислот (а еще белков) в пространстве, занимается целая «омиковая наука», называемая структуромикой. Проблемы предсказания термодинамически устойчивых структур, особенно таких больших, какими являются обозначенные выше биополимеры, не решить без операций программного отсеивания и сопоставления больших массивов данных, поэтому задачи определения реальных форм ДНК, РНК и белков относят к биоинформатическим. Подробнее есть на «Биомолекуле»: «Биоинформатика в мире РНК-структур» [32].
Наконец, представим проекцию вторичной структуры части РНК вируса гриппа A, ответственную за кодирование одной из субъединиц вирусной полимеразы (так называемой субъединицы PB-1). В ней содержится такое огромное число нуклеотидов (более 2000), что ее вид, даже представленный в виде проекции, здесь бы просто не уместился. Очевидно, что вероятность присоединения дезоксирибозимного «макроса» к данному участку в реальных условиях крайне мала.
Иными словами, просто функции распознавания целевого фрагмента и ее расщепления уже недостаточно для эффективной работы системы и выполнения поставленных перед ней задач. Необходимо включать в ее программный код и другие функции. Первой из таких напрашивается расплетение вторичной структуры РНК вблизи целевого участка для лучшего распознавания и связывания. Второй — стабилизация комплекса РНК—дезоксирибозим. Для повышения эффективности также можно добавить в систему еще несколько дезоксирибозимов, нацеленных на другие, расположенные поблизости участки.
Итоговый комплекс молекул, выполняющих различные функции и соединенных между собой по принципу комплементарности, уже можно будет называть машиной, которая запрограммирована на выполнение множества операций в зависимости от окружающих условий без контроля со стороны оператора. Это полностью отвечает понятию «робот», и потому подобные сложные конструкции и называются ДНК-нанороботами, и подобный термин уже появлялся на сайте [25].
Основы для программных кодов таких ДНК-нанороботов, направленных на различные задачи, приведены на рисунках 12 и 13.
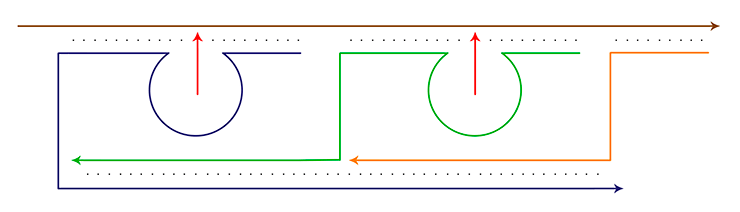
Рисунок 12. Пример наноробота, направленного на расщепление РНК вируса гриппа A и выполняющего следующие функции: распознавание вирусной РНК (коричневый фрагмент) и присоединение к ней, расщепление в двух местах, благодаря дезоксирибозимным участкам (синий и зеленый фрагменты), расплетение и стабилизация вирусной РНК (оранжевый фрагмент). Для упрощения схемы нуклеотидный код каждой цепи не указан.
Рисунок 13. Наноробот, направленный на обнаружение генетической информации палочки Коха в пробе. В основе робота лежит двусоставный дезоксирибозим (зеленый) [33], [34], который расщепляет молекулярный маяк только в присутствии последовательности, принадлежащей туберкулезной микобактерии (коричневый), вызывая флуоресцентный сигнал. Сложная конструкция основы (синий) обеспечивает роботу бóльшую стабильность, а также выполняет функции расплетения вторичной структуры целевой последовательности и концентрирует вокруг себя молекулы маяков. Для упрощения схемы нуклеотидный код каждой цепи не указан.
Заключение
Подводя итог всему вышесказанному, хотелось бы отметить, что мир молекул РНК и ДНК, в отрыве от привычной для нас функции «передачи информации», таит в себе множество интересных возможностей и неожиданных проявлений. Более того, многогранность этих возможностей позволяет рассматривать их с совершенно различных сторон — в частности, как и было представлено в этой статье, работа механизмов на основе нуклеиновых кислот может быть представлена даже в виде функционирования реально существующего языка программирования.
Наличие подобных ракурсов, не лишенных изрядной доли смысла, возможно, станет интересным для людей самых различных профессий и специальностей, что станет причиной вовлеченности в изучение основы нашего существования все большего и большего числа людей.
В самом начале данной статьи уже упоминалось, что подобный взгляд, да и сам термин «ДНК-программирование» являются неотъемлемыми составляющими одной большой прикладной науки, носящей название синтетической биологии. Изучить живое, «взломать» его программный код, чтобы понять, как это живое работает, и на основе полученных знаний создать нечто новое — задача целого ряда дисциплин и уровней, начиная от организменного и заканчивая молекулярным и даже атомным. Наука не стоит на месте и, возможно, уже в самом ближайшем времени мы сможем «перепрограммировать» самих себя.